創新時間序列分析方法用以重建大型系統交互作用網絡
張俊偉、謝志豪
由臺大海洋所謝志豪教授與國家理論科學中心張俊偉博士所帶領的國際研究團隊研發出利用時間序列資料來重建大型交互作用網絡的新方法。這項研究成果於2021年10月發表在頂尖生態學期刊《生態學通訊》(Ecology Letters)。這個統計新方法克服了過去在自然觀測的條件下,重建大型交互作用網絡的挑戰。團隊所提出的分析工具將成為探索自然界中交互作用網絡之一大利器。
以實際觀測資料來重建自然系統中生物之間的交互作用與其所形成的複雜網絡,是個重要卻極具挑戰的工作,特別是在於重建高物種多樣性和物種交互作用的生態系統上,例如具有高度生物多樣性的微生物群聚、珊瑚礁、或是熱帶雨林生態系統。過往研究所提出的網絡重建方法僅能量化小型系統內的交互作用 (例如:物種數少於20種的情況),而且難以解決網絡隨時間變化之難題。為解決此一難題,該研究團隊提出了一個全新的方法:多視點距離正則化S映射 (Multiview distance regularized S-map; MDR S-map)。這個方法克服了量化大型交互作用網絡時所面臨的維數災難議題(亦即,以有限的資料推估大量參數的困境;Curse of dimensionality)。這個新方法成功地重建了數個理論模型背後的大型交互作用網絡–包含了數以百計的物種和它們之間隨時間改變之交互作用。
研究團隊更成功地將這樣的方法應用在真實的海洋微生物群聚,重建了細菌種類之間的交互作用網絡,並且得以從這些網絡中辨識出扮演關鍵角色的菌種。而量化的網絡結構也讓科學家得以檢驗一個高度複雜的動態系統,並找出影響系統動態穩定性背後的複雜機制。
這個新方法滿足了科學界對於重建大型交互作用網絡的強大需求。特別是在生態學領域,生態學家總是竭力探索存在於諸多生物物種之間各式各樣的交互作用(例如:掠食、競爭、合作等…)。藉由這個新方法,科學家僅需要對於有興趣的系統進行定時的量化觀測,就可了解甚至於預測複雜之生物交互作用網絡。研究團隊認為這個新方法不只可以被應用在生態系統,甚至也可以應用在醫學與其他領域(例如:癌細胞的蛋白質交互作用網絡如何因化療而改變,神經網絡如何隨時間變異),也可以用來探討非生物系統,例如經濟系統或是傳染病網絡,來解析各種類型的複雜交互作用網絡。
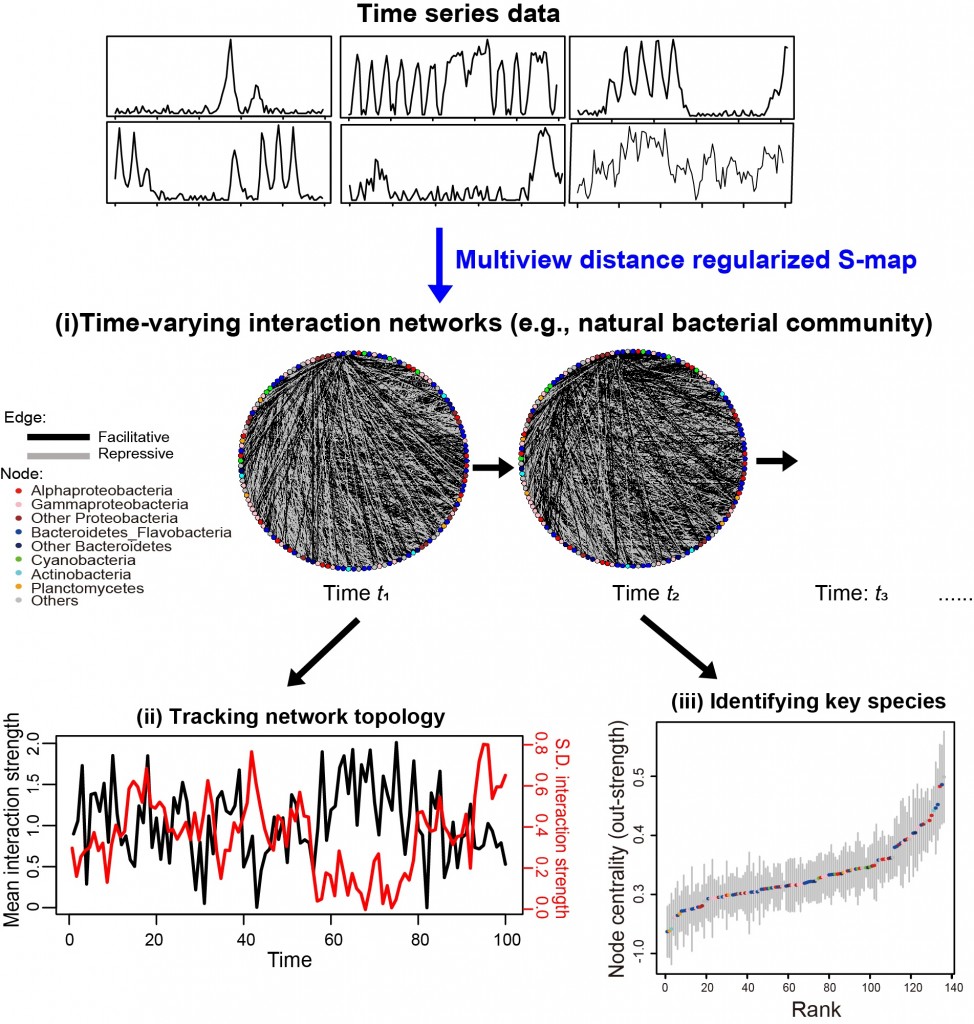
圖1、透過所研發之新方法MDR S-map,分析眾多物種(網絡節點)的時間序列資料,得以重建物種之間的複雜交互作用網絡,且能追蹤這些網絡結構如何隨時間變化。該研究分析了採集自美國東海岸的野外細菌群聚,並且重建了135個細菌物種之間的交互作用網絡。藉由分析重建後的網絡(i),量化並追蹤重要的網絡拓樸結構如何隨時間變化(ii),也透過分析物種間之交互作用來決定網絡中扮演關鍵角色的物種(iii)。
延伸閱讀
Chang, C. W., T. Miki, M. Ushio, P. J. Ke, H. P. Lu, F. K. Shiah, C. h. Hsieh. (2021) Reconstructing large interaction networks from empirical time series data. Ecology Letters. https://doi.org/10.1111/ele.13897